R para Entomologistas: nível básico
CAPÍTULO 3
Análise de variáveis contínuas coletadas em experimentos com esquemas fatoriais
José Bruno Malaquias, Tatiane Caroline Grella, Jéssica Karina da Silva Pachú
https://doi.org/10.4322/mp.2020-12.c3
Resumo
Nesta seção, estamos expondo um exemplo de análise de banco de dados hipotético que foi delineado em blocos ao acaso e com estrutura de tratamento em esquema fatorial 2 x 2. Como a variável hipotética é contínua, então novamente exploramos algumas linhas de comando que são essenciais para testes de normalidade e homogeneidade de variâncias, para posterior análise de variância e testes de comparação de médias. Apresentamos todas as linhas de comando com um passo a passo de forma comentada e detalhada. Utilizaremos os seguintes pacotes: readxl, MASS e ExpDes.pt. Os comandos a serem executados no R estão sendo apresentados com a cor azul.
Palavras-chave: análise fatorial; ANOVA; dois fatores; variável contínua; desdobramento; interação.
1. Importação do banco de dados
Após fazer o download dos arquivos utilizados neste capítulo ao clicar aqui, é necessário verificar em qual local do computador (diretório) o arquivo que será analisado se encontra.
Para isso, vamos usar o comando getwd(), que irá verificar em qual diretório você está trabalhando: getwd()
Caso seja necessário alterar o diretório, basta seguir os passos:
Session -> Set working Directory -> Choose Directory
Após escolher um diretório é possivel ver quais os arquivos existem no mesmo, para isso, usamos a função: list.files() que mostrará a lista de arquivos dentro do seu diretório.
Após a escolha do diretório, vamos ler o arquivo que será analisado.
Para ler o arquivo em excel, nós iremos precisar do pacote readxl [1].
Uma forma elegante de carregar o pacote é utilizar a linha de comando (essa linha também funciona caso o pacote não esteja instalado, pois automaticamente a instalação será realizada):
if (!require("readxl")) install.packages("readxl"); require(readxl)
O sinal de exclamação indica negação, então a linha de comando acima é traduzida como: "caso o pacote necessário (readxl) nao esteja instalado, instale o pacote, e em seguida carregue-o"
Vamos agora explorar uma outra forma de ler o banco de dados, mas utilizando o mesmo pacote. Primeiro vamos colocar o nosso path para leitura do arquivo, veja que atribui o meu path com o nome MinhaPastaEArquivo (cada um pode atribuir o nome que desejar):
MinhaPastaEArquivo <- "C:/Users/Bruno/Google Drive/Grupo de Discussão/Grupo - Linguagem R/Material-01082020/BD1-Anovatwoway.xlsx"
Veja que parte do path é pessoal: "C:/Users/Bruno/Google Drive/Grupo de Discussão/Grupo - Linguagem R/Material-01082020
É possível extrair essa parte, apenas rodando o comando getwd(), copiando e colando o que aparecer.
A outra parte extraímos do resultado de list.files()
Para mostrar os nomes das planilhas disponíveis, é necessário executar a seguinte linha de comando:
excel_sheets(path=MinhaPastaEArquivo)
Vamos utilizar agora a função read_excel para ler o arquivo e especificar a planilha que temos interesse.
df <-read_excel(path=MinhaPastaEArquivo, sheet = "Planilha1")
Veja que estamos atribuindo o nome do banco de dados agora de df
head(df): para ler o cabeçalho do dataframe.
View(df): para ver o banco de dados em outra janela
attach(df): para enviar o banco de dados para a memória
2. Testes de pressuposição da ANOVA
Para testar a homogeneidade de variâncias, iremos utilizar o teste de Bartlett, com a função bartlett.test
Vresp: é a variável resposta de interesse, ou variável dependente
Fator1: é o fator 1, ou a variável independente 1
Fator2: é o fator 1, ou a variável independente 2
Bloco: é o fator 1, ou a variável independente 2
bartlett.test(df$Vresp, df$Fator1) |
teste de Bartlett para um estudo conduzido em DIC - com apenas um fator. |
bartlett.test(df$Vresp, df$Fator1, df$Bloco) |
teste de Bartlett para um estudo conduzido em DBC - com apenas um fator. |
bartlett.test(df$Vresp, df$Fator1, df$Fator2) |
teste de Bartlett para um estudo conduzido em DIC com esquema fatorial (2 fatores). |
bartlett.test(df$Vresp, df$Fator1, df$Fator2, df$Bloco) |
teste de Bartlett para um estudo conduzido em DBC com esquema fatorial (2 fatores). |
Teste de Bartlett: Se o p-value for superior a 0.05, as variâncias são homogêneas.
Teste de Shapiro: shapiro.test
Se o p-value for superior a 0.05, há normalidade dos dados.
3. ANOVA com dois fatores (two-way ANOVA)
Esse é o modelo que iremos trabalhar para two way-ANOVA:
Modelofatorial<-aov(ALT~FAT1*FAT2+BLOC, data=df)
Veja que usamos a função aov para rodar a ANOVA.
ALT: é a variável resposta.
FAT1: é o fator 1.
FAT2: é o fator 2.
BLOC: é o fator bloco.
Essas designações ALT, FAT1, FAT2 e BLOC são provenientes do seguinte banco de dados:
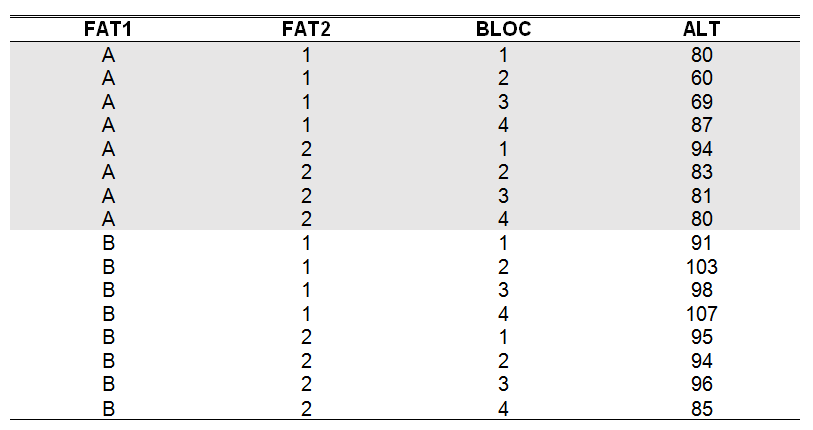
summary(Modelofatorial): mostra o resumo da ANOVA
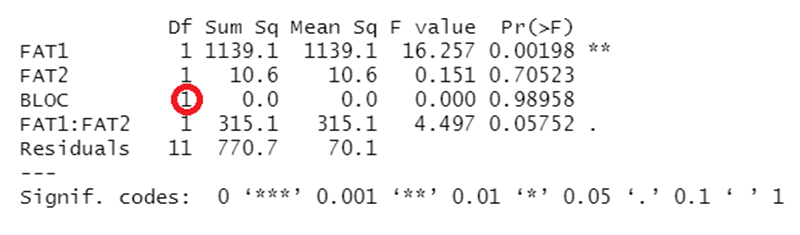
Veja que essa análise contém um erro. O erro desse output está no número de graus de liberdade para o fator BLOC (que corresponde a blocos). O número correto seria 3. A fórmula de graus de liberdade é n-1, nesse caso temos 4 blocos, que perfazeria BLOC= 3.
Antes de seguir com a análise precisamos perguntar se é fator, porque temos variáveis dependentes e independentes, quando elas são dependentes precisa ser fator, para isso usamos o comando:
is.factor(df$FAT1)
is.factor(df$FAT2)
is.factor(df$BLOC)
Quando as variáveis são independentes é necessário converter, para isso precisamos utilizar a função as.factor e atualizar cada fator convertido
df$FAT1<-as.factor(df$FAT1)
df$FAT2<-as.factor(df$FAT2)
df$BLOC<-as.factor(df$BLOC)
ATENÇÃO: não faça esse procedimento de conversão em fator para as suas variáveis dependentes (variáveis respostas).
Após a conversão das variáveis independentes em fator, nós precisamos testar novamente se as variáveis são fatores:
is.factor(df$FAT1)
is.factor(df$FAT2)
is.factor(df$BLOC)
o resultado deverá ser TRUE
Agora que as suas variáveis independentes são fatores, você pode rodar a two-way ANOVA.
Modelofatorial<-aov(ALT~FAT1*FAT2+BLOC, data=df)
ummary(Modelofatorial)
Perceba que o número de graus de liberdade do fator BLOC agora está correto!!
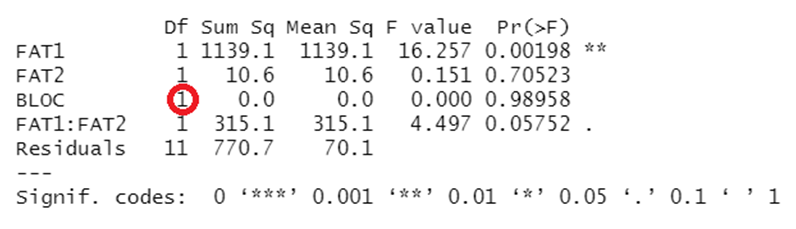
Agora poderemos aplicar os testes de comparações de médias. Para isto, vamos utilizar a função fat2.dbc, vamos carregar o pacote com a nossa forma elegante:
if(!require("ExpDes.pt")) install.packages("ExpDes.pt"); require(ExpDes.pt)
fat2.dbc(FAT1, FAT2, BLOC, ALT, quali = c(TRUE, TRUE), mcomp = "tukey",
fac.names = c("FATOR 1", "FATOR 2"), sigT = 0.05, sigF = 0.05)
Como a interação não foi significativa (podemos observar isso na linha "FAT1:FAT2") serão analisados apenas os efeitos simples.
4. Outros exemplos
Para acessar o script e banco de dados, clique aqui. Vamos escolher o diretório no qual queremos trabalhar:
Session -> Set working Directory -> Choose Directory
Abra a pasta que contém o seu banco de dados.
Vamos usar o comando list.files() para visualizarmos os nomes de todos os arquivos que contém na pasta selecionada.
Quando seu banco de dados está no formato do Excel é necessário rodar o comando
df<-read_excel("BD.xlsx", sheet = 1), onde:
![]() df é o nome dado ao dataframe (você pode colocar o nome que quiser);
df é o nome dado ao dataframe (você pode colocar o nome que quiser);
![]() read_excel é o comando para ler o arquivo do Excel
read_excel é o comando para ler o arquivo do Excel
![]() BD. é o nome do seu arquivo dentro da pasta selecionada
BD. é o nome do seu arquivo dentro da pasta selecionada
![]() xlsx é a extensão do arquivo com o qual você está trabalhando
xlsx é a extensão do arquivo com o qual você está trabalhando
![]() sheet = 1 faz referência a qual aba do seu arquivo do Excel quer analisar
sheet = 1 faz referência a qual aba do seu arquivo do Excel quer analisar
Para ver o cabeçalho do banco de dados a função utilizada é: head(df, n=2) neste caso, em específico, será visualizada apenas 2 linhas do seu banco de dados.
Antes de realizarmos os testes de normalidade e homogeneidade é necessário verificar se as variáveis independentes são fatores.
Neste caso, as variáveis são: Ins, Fung e Bloco
Ins |
Fung |
Bloco |
A |
X |
1 |
A |
X |
2 |
A |
X |
3 |
A |
X |
4 |
B |
X |
1 |
B |
X |
2 |
B |
X |
3 |
Para realizar a verificação é necessário utilizar os comandos:
is.factor(df$Ins)
is.factor(df$Fung)
is.factor(df$Bloco)
A resposta para a verificação precisa ser: TRUE
Caso a resposta de: FALSE, é necessário converter as variáveis independentes em fatores, para isso utilizamos a função "as.factor":
df$Ins<-as.factor(df$Ins)
df$Fung<-as.factor(df$Fung)
df$Bloco<-as.factor(df$Bloco)
em seguida é necessário verificar se a conversão foi bem sucedida:
is.factor(df$Ins)
is.factor(df$Fung)
is.factor(df$Bloco)
Em seguida é necessário selecionar qual variável deseja analisar
df$VRESP<-df$D
Neste caso, trabalharemos com a variável "D", proveniente do banco de dado.
Atenção: o nome da variável deve ser idêntico ao escrito no banco de dado.
Depois de selecionarmos a variável que queremos analisar, podemos realizar os testes de normalidade e homogeneidade:
Para normalidade usamos o comando:
shapiro.test(df$VRESP)
Shapiro-Wilk normality test
data: df$VRESP
W = 0.95084, p-value = 0.5031
Para que os dados sejam considerados normais, o valor de "p" tem que ser maior que 0.05. Dessa forma podemos verificar que a análise em questão passou no teste de normalidade, pois p=0.5031.
Para homogeneidade usamos o comando:
bartlett.test(df$VRESP, df$Ins, df$Fung, df$Bloco)
Bartlett test of homogeneity of variances
data: df$VRESP and df$Ins
Bartlett's K-squared = 2.3704, df = 1, p-value = 0.1237
Para que os dados sejam considerados homogêneos, o valor de "p" tem que ser maior que 0.05. Dessa forma podemos verificar que a análise em questão passou no teste de homogeneidade, pois p=0.1237.
OBS: caso os dados não atendam à normalidade e homogeneidade é necessário transforma-los, verifique como fazer isso mais a baixo.
Em seguida podemos rodar a ANOVA, para isso utilizaremos a função "aov":
Modelofatorial<-aov(VRESP~Ins*Fung+Bloco, data=df)
summary(Modelofatorial)
Em seguida fazemos um Summary para verificar o resultado do teste a cima:
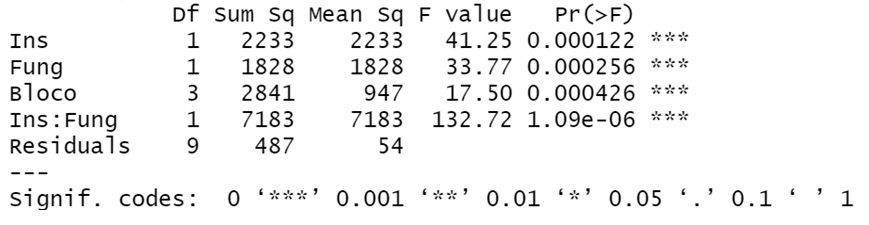
Através do teste realizado podemos observar que existe diferença significativa nos grupos quando analisados separadamente e também na interação inseticida versus fungicida.
OBS: quando a interação não for significativa, não é necessário fazer desdobramento da interação inseticida versus fungicida.
Quando ocorre diferença significativa podemos rodar o teste de Tukey, o qual faz comparações múltiplas, para isso é necessário instalar o pacote "ExpDes" [2], utilizando o comando:
if(!require("ExpDes.pt")) install.packages("ExpDes.pt"); require(ExpDes.pt)
Esse comando verifica se você possui o pacote necessário e caso não possua, ele baixa e instala esse pacote.
Após a instalação do pacote, podemos rodar o teste de Tukey, com o seguinte comando:

Esse comando já especifica que a significância dos resultados é de 95%
Como resultado do teste de Tukey, obtemos os desdobramentos:

![]() Letras diferentes indicam que houve diferença significativa entre os tratamentos.Para apresentarmos os resultados finais nos artigos, utilizamos essas letras, mas paraobtermos a média juntamente com uma medida de variabilidade desvio ou erro padrão que devemos apresentar é necessário instalar o pacote "Rmisc" e rodar um último co-mando:
Letras diferentes indicam que houve diferença significativa entre os tratamentos.Para apresentarmos os resultados finais nos artigos, utilizamos essas letras, mas paraobtermos a média juntamente com uma medida de variabilidade desvio ou erro padrão que devemos apresentar é necessário instalar o pacote "Rmisc" e rodar um último co-mando:
if (!require("Rmisc")) install.packages("Rmisc"); require(Rmisc)
summarySE(df, measurevar = "VRESP", groupvars = c("Ins", "Fung"))
Obtemos como resultado:

Um exemplo de tabela que pode ser construída a partir dos resultados obtidos é:

Letras maiúsculas comparam colunas.
Letras minúsculas comparam linhas.
5. Caso que necessita de transformação
Clique aqui para acessar o banco de dados e script.
Vamos ler o banco de dados:
df<-read_excel("BD (1).xlsx", sheet = 1)
head(df, n=2)
Vamos testar as pressuposições:
shapiro.test(df$VRESP)

bartlett.test(df$VRESP, df$Ins, df$Fung, df$Bloco)

Perceba que os dados não atendarem às pressuposições de normalidade e homogeneidade de variâncias (p < 0.05). Portanto, é necessário transformá-los, para isso, precisaremos do pacote MASS [3] e usaremos o comando:
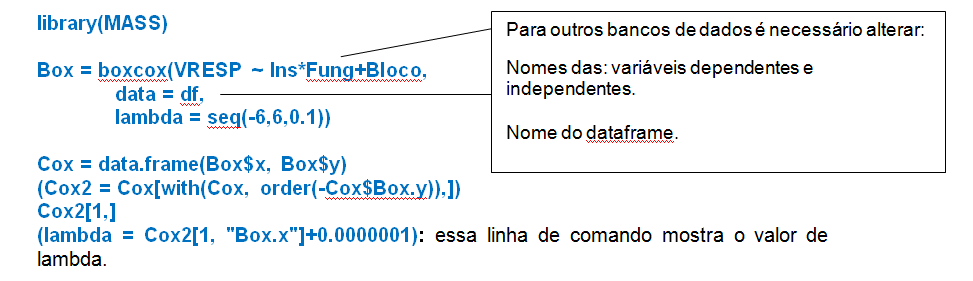
(df$VRESP_box = (df$VRESP ^ lambda - 1)/lambda): aqui inserimos a fórmula de Box-Cox [5].
Após a transformação devemos repetir os testes "Shapiro" e "Bartlett" para verificar se os dados atendem as pressuposições de normalidade e homogeneidade (SE ATENTAR A FORMA DE ESCREVER O COMANDO, POIS AGORA OS DADOS ESTÃO TRANSFORMADOS, ENTÀO É NECESSÁRIO ACRESCENTAR "box").
shapiro.test(df$VRESP_box)
bartlett.test(df$VRESP_box, df$Ins, df$Fung, df$Bloco)
Após isso vamos rodar a análise de variância:
if(!require("ExpDes.pt")) install.packages("ExpDes.pt"); require(ExpDes.pt)
fat2.dbc(Ins, Fung, Bloco, VRESP_box, quali = c(TRUE, TRUE), mcomp = "tukey",
fac.names = c("Inseticida", "Fungicida"), sigT = 0.05, sigF = 0.05)
if (!require("Rmisc")) install.packages("Rmisc"); require(Rmisc)
summarySE(df, measurevar = "VRESP", groupvars = c("Ins", "Fung"))
Quando é necessário fazer a transformação dos dados, devemos nos atentar para os valores de médias que colocamos na tabela dos resultados finais, pois elas não devem ser dos dados transformados e sim aqueles originais, os quais são obtidos na análise descritiva acima com a função summarySE do pacote Rmisc [4].
6. Referências dos pacotes utilizados
[1] Wicham H., Bryan J. readxl: Read Excel Files. R package version 1.3.1. 2019. https://CRAN.R-project.org/package=readxl
[2] Ferreira E.B., Cavalcanti P.P., Nogueira D.A. ExpDes.pt: Pacote Experimental Designs (Portuguese). R package version 1.2.0. 2018. https://CRAN.R-project.org/package=ExpDes.pt
[3] Venables W.N., Ripley B.D. Modern Applied Statistics with S. Fourth Edition. Springer, New York. 2002.
[4] Hope R.M. Rmisc: Rmisc: Ryan Miscellaneous. R package version 1.5. https://CRAN.R-project.org/package=Rmisc. 2013.
[5] Box G., Cox DR. An analysis of transformations. Journal of the Royal Society, 26: 211-252. 1964.
7. Referências recomendadas
Crawley, M.J. The R book. John Wiley & Sons, 2012.
Matloff Norman. The art of R programming: A tour of statistical software design. No Starch Press, 2011.
Peternelli L.A., Mello M.P. Conhecendo o R: uma visão estatística. Viçosa: UFV, v. 1, 2011.
Autores
José Bruno Malaquias, Instituto de Biociências - Câmpus de Botucatu. R. Prof. Dr. Antônio Celso Wagner Zanin, 250 - Distrito de Rubião Junior - Botucatu/SP, Brasil - CEP 18618-689
Tatiane Caroline Grella - Biologia Celular e Molecular na Universidade Estadual Paulista "Júlio de Mesquita Filho" - Laboratório de Ecotoxicologia e Conservação de Abelhas (LECA) - Avenida 24 A,1515 - Bela Vista - CEP 13506-900 - Rio Claro/SP, Brasil
Jéssica Karina da Silva Pachú, Departamento de Entomologia e Acarologia - LEA Avenida Pádua Dias, 11 - CEP 13418-900 - Piracicaba/SP, Brasil
* Autor para correspondência: malaquias.josebruno@gmail.com
