R para Entomologistas: nível básico
CAPÍTULO 2
Análise descritiva e testes de pressuposições para análise de variância
Tatiane Caroline Grella, José Bruno Malaquias, Jéssica Karina da Silva Pachú
https://doi.org/10.4322/mp.2020-12.c2
Resumo
Neste capítulo, descrevemos de forma simples e objetiva elementos essenciais para análise descritiva no R por meio de boxplot e também exploramos algumas linhas de comando que são essenciais para testes de normalidade e homogeneidade de variâncias, para posterior análise de variância e testes de comparação de duas médias, no caso teste t, e comparação múltipla de médias - teste de Tukey. Todas as linhas de comando são apresentadas com um passo a passo de forma comentada e detalhada. Utilizaremos os seguintes pacotes: readxl e ExpDes.pt. Os comandos a serem executados no R estão sendo apresentados com a cor azul.
Palavras-chave: boxplot; normalidade; homocedasticidade; variáveis contínuas; testes de comparação; ANOVA.
1. Leitura de arquivos no R
Para acessar o banco de dados e o script utilizados como exemplo neste capítulo clique aqui.
Para iniciar a análise é necessário fazer o download do banco de dados e script (disponibilizados a cima) e em seguida verificar em qual local do computador (diretório) o arquivo que será analisado se encontra.
Para isso, vamos usar o comando getwd, que irá verificar em qual diretório você está trabalhando: getwd()
Caso seja necessário alterar o diretório, basta seguir os passos:
Session -> Set working Directory -> Choose Directory
Após escolher um diretório é possivel ver quais os arquivos existem no mesmo, para isso, usamos a função: list.files() que mostrará a lista de arquivos dentro do seu diretório.
Após a escolha do diretório, vamos ler o arquivo que será analisado.
Para ler o arquivo em excel, nós iremos precisar do pacote: readxl [1].
Uma forma elegante de carregar o pacote é utilizar a seguinte linha de comando (essa linha também funciona caso o pacote não esteja instalado, pois automaticamente a instalação será realizada):
if(!require("readxl")) install.packages("readxl"); require(readxl)
O sinal de exclamação indica negação, então a linha de comando acima é traduzida como: "caso o pacote necessário (readxl) nao esteja instalado, instale o pacote, e em seguida carregue-o".
2. Análise descritiva com box plot
Depois de carregar o pacote, precisamos ler o arquivo, para isso usaremos a linha de comando:
df<-read_excel("BD1.xls ", sheet = 1)
![]() df é o nome dado ao dataframe (você pode colocar o nome que desejar);
df é o nome dado ao dataframe (você pode colocar o nome que desejar);
![]() read_excel é o comando para ler o arquivo do Excel
read_excel é o comando para ler o arquivo do Excel
![]() BD1 é o nome do seu arquivo dentro da pasta selecionada
BD1 é o nome do seu arquivo dentro da pasta selecionada
![]() xls é a extensão do arquivo com o qual você está trabalhando
xls é a extensão do arquivo com o qual você está trabalhando
![]() sheet = 1 faz referência a qual aba do seu arquivo do Excel quer analisar
sheet = 1 faz referência a qual aba do seu arquivo do Excel quer analisar
Vamos ler o Arquivo: BD1.xls.
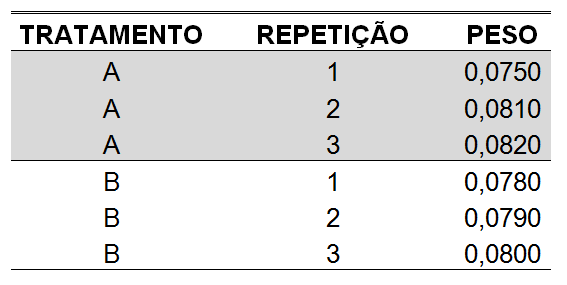
Nesse exemplo didático foi analisado o efeito de dois tratamentos no peso dos insetos (Tabela 1). O delineamento experimental foi inteiramente casualizado e com apenas 3 repetições.
Tabela 1 - Peso (g) de uma espécie hipotética de inseto submetida a dois tratamentos
Para ver o cabeçalho usamos a função: head(df)
Para ver o banco de dados, usamos a função: View(df)
Para expressar os dados em um boxplot e construi-lo, usamos a função:
boxplot(PESO~TRATAMENTO, data=df)
Após a construção é possivel alterar diversos itens, como:
![]() nome nos eixos, usando o comando:
nome nos eixos, usando o comando:
boxplot(PESO~TRATAMENTO, data=df,
xlab = "Tratamentos",
ylab = "Matéria seca (kg)")
Neste exemplo os nomes do eixos são "Tratamentos" "Matéria seca (kg)"
![]() nome nos eixos e com limite inferior e superior.
nome nos eixos e com limite inferior e superior.
boxplot(PESO~TRATAMENTO, data=df,
xlab = "Tratamentos",
ylab = "Matéria seca (kg)"
, ylim = c(0.07,0.085))
![]() nome nos eixos e com limite inferior e superior e adicionando a média dentro do Boxplot.
nome nos eixos e com limite inferior e superior e adicionando a média dentro do Boxplot.
boxplot(PESO~TRATAMENTO, data=df, col= "white",
xlab= "Variedades", ylab= "Matéria Seca (Kg)", ylim = c(0.07,0.085))
points(1:nlevels(TRATAMENTO), tapply(PESO, TRATAMENTO, mean), data=df)
abline(mean(TRATAMENTO), data=df)
Para exportar a figura no formato desejado (uma sugestão é o formato tiff com 300 dpi, que geralmente é o formato solicitado pelos periódicos).
Para alterar os tamanhos, utilize: cex.main (título), cex.lab (rótulos) e cex.axis (tamanho dos eixos).
tiff("Figura_BoxPlot_.tiff", width=12, height=8, units="in", res=300)
boxplot(PESO~TRATAMENTO, data=df, col= "white",
xlab= "Variedades", ylab= "Matéria Seca (Kg)",
ylim = c(0.07,0.085),
cex.main=1.5, cex.lab=1.5, cex.axis=1.5)
dev.off()
OBS: observe o seu diretório, pois a figura será diretamente exportada para essa pasta.
Para escolher o diretório utilize Control + Shift + H ou simplesmente use getwd()
A Figura 1 é o boxplot exportado. Cada seta expressa um parâmetro da análise descritiva, ou seja, os valores mínimo, máximo e quantis (1°, 2° e 3°).
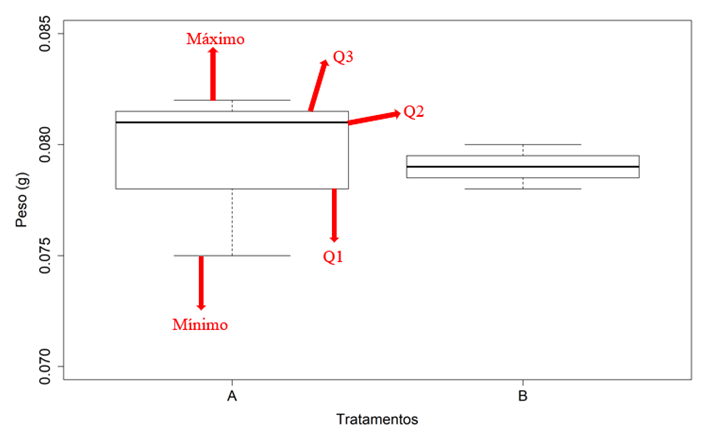
Figura 1 - Boxplot representando o efeito de dois tratamentos no peso (g) de uma espécie hipotética de inseto. Q1: primeiro quantil. Q2: segundo quantil ou mediana. Q3: terceiro quantil.
Com a utilização da linha de comanda abaixo, será possível visualizar os mesmos valores expressos no boxplot, ou seja, valor mínimo (Min), primeiro quantil (1st Qu.), mediana (Median), média (Mean), terceiro quantil (3rd Qu.) e máximo (Max), portanto, será demonstrado um resumo da análise descritiva do peso (g) de uma espécie hipotética de inseto submetida a cada um dos dois tratamentos.
tapply(df$PESO, df$TRATAMENTO, summary)

3. Testando as pressuposições do modelo da ANOVA
Vamos utilizar o mesmo banco de dados que foi utilizado anteriormente, então leia o arquivo: BD1.xls (Tabela 1) utilizando o script que está disponível ao clicar aqui. Conforme exposto na Tabela 1 e comentado anteriormente, nesse exemplo didático foi analisado o efeito de dois tratamentos no peso dos insetos. O delineamento experimental foi inteiramente casualizado e com apenas 3 repetições.
Carregue o pacote readxl, para ler o arquivo do Excel:
require(readxl) caso tenha dúvidas em relação ao carregamento do pacote e instalação, veja o tópico "Leitura de arquivos no R".
Leia o banco de dados: df<- read_excel("BD1.xlsx", sheet = 1)
Ver o cabeçalho: head(df)
Ver o banco de dados: View(df)
Antes de realizar a análise ANOVA, é necessário testar a normalidade e a homogeneidade das variâncias.
Para testar a normalidade, utilizamos o teste de Shapiro Wilk, usando:
shapiro.test(df$PESO)
Teste de Shapiro: Se o p-value for superior a 0,05 (5%), há normalidade dos dados.
Para testar a homogeneidade, em um estudo conduzido em DIC - com apenas um fator, utilizamos o teste de Bartlett, usando:
bartlett.test(df$PESO, df$TRATAMENTO)
Teste de Bartlett: Se o p-value for superior a 0.05, as variâncias são homogêneas.
OBS: caso seus dados sejam variáveis contínuas e não atendam à normalidade e/ou homogeneidade serão necessárias transformações.
4. Teste T
Vamos continuar utilizando o mesmo banco de dados que foi utilizado anteriormente, mas com alteração do script que podem ser acessados clicando aqui, então leia o Arquivo: BD1.xls (Tabela 1). Como são apenas dois tratamentos, iremos aplicar um teste t. Mas antes disso, precisaremos testar as pressuposições de normalidade e homogeneidade de variâncias, para maiores detalhes veja o tópico: "Testando as Pressuposições do Modelo da Anova".
Carregue o pacote readxl, para ler o arquivo do Excel:
require(readxl) caso tenha dúvidas em relação ao carregamento do pacote e instalação, veja o tópico "Leitura de arquivos no R".
Leia o banco de dados: df<- read_excel("BD1.xlsx", sheet = 1)
Ver o cabeçalho: head(df)
Ver o banco de dados: View(df)
Teste de Shapiro Wilk: shapiro.test(df$PESO)
Teste de Bartlett para um estudo conduzido em DIC - - com apenas um fator: bartlett.test(df$PESO, df$TRATAMENTO)
Para aplicar o teste t, utilize a função t.test.
![]() Peso é a variável resposta;
Peso é a variável resposta;
![]() Tratamento é a variável independente.
Tratamento é a variável independente.
Pode-se utilizar as opções: "two.sided", "less" ou "greater".
t.test(PESO ~ TRATAMENTO, data = df, alternative = "two.sided")
5. Teste de Tukey
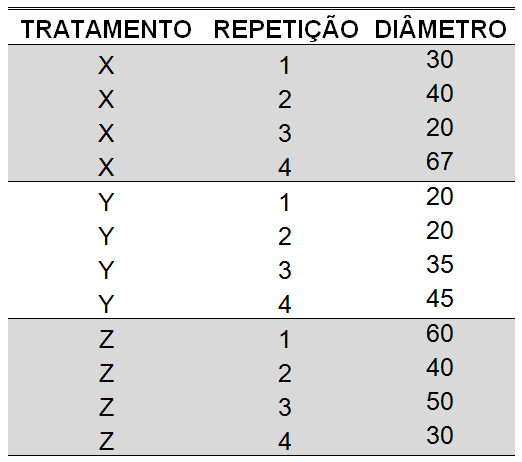
Vamos utilizar o banco de dados presente no Arquivo: Anova1.0.xlsx (Tabela 2). O delineamento experimental foi inteiramente casualizado e com 4 repetições. Como são mais de dois tratamentos, iremos aplicar um teste de comparações múltiplas, nesse caso o teste de Tukey. Lembre-se, de testar as pressuposições de normalidade e homogeneidade de variâncias, para maiores detalhes veja o tópico: Testando as Pressuposições do Modelo da Anova.
Tabela 2 - Diâmetro (mm) do pronotum de uma espécie hipotética de inseto submetida a três tratamentos
Carregue o pacote readxl, para ler o arquivo do Excel:
require(readxl) caso tenha dúvidas em relação ao carregamento do pacote e instalação, veja o tópico "Leitura de arquivos no R".
Leia o banco de dados: df<- read_excel("BD1.xlsx", sheet = 1)
Ver o cabeçalho: head(df)
Ver o banco de dados: View(df)
Teste de Shapiro Wilk: shapiro.test(df$Diametro)
Teste de Bartlett para um estudo conduzido em DIC - com apenas um fator: bartlett.test(df$Diametro, df$Tratamento)
Para essa análise vamos utilizar o pacote ExpDes.pt [2]: require(ExpDes.pt)
Para enviar os comandos digitados (data frame) para memória: attach(df)
O ensaio foi conduzido no delineamento "dic", por isso vamos utilizar a função dic
#em mcomp, escolha o método: "tukey"
dic(Tratamento, Diametro, quali = TRUE, mcomp = "tukey", nl=FALSE, sigT = 0.05, sigF = 0.05)
Esse é o resultado da análise:

A partir da análise, percebe-se que não existem evidências de diferenças entre os tratamentos. Caso queira apresentar os resultados usando letras, basta atribuir a mesma letra para as 3 médias - embora isto seja considerado redundante.
6. Referências dos pacotes utilizados
[1] WICKHAM H., BRYAN J. readxl: Read Excel Files. R package version 1.3.1. 2019. https://CRAN.R-project.org/package=readxl
[2] FERREIRA E.B., CAVALCANTI P.P., NOGUEIRA D.A. ExpDes.pt: Pacote Experimental Designs (Portuguese). R package version 1.2.0. 2018. https://CRAN.R-project.org/package=ExpDes.pt
7. Referências recomendadas
CRAWLEY, Michael J. The R book. John Wiley & Sons, 2012.
MATLOFF, Norman. The art of R programming: A tour of statistical software design. No Starch Press, 2011.
PETERNELLI, Luiz Alexandre; MELLO, MP de. Conhecendo o R: uma visão estatística. Viçosa: UFV, v. 1, 2011.
VENABLES W.N., RIPLEY B. D. Modern Applied Statistics with S. Fourth Edition. Springer, New York. 2002.
Autores
Tatiane Caroline Grella - Biologia Celular e Molecular na Universidade Estadual Paulista "Júlio de Mesquita Filho" - Laboratório de Ecotoxicologia e Conservação de Abelhas (LECA) - Avenida 24 A,1515 - Bela Vista - CEP 13506-900 - Rio Claro/SP, Brasil
José Bruno Malaquias, Instituto de Biociências - Câmpus de Botucatu. R. Prof. Dr. Antônio Celso Wagner Zanin, 250 - Distrito de Rubião Junior - Botucatu/SP, Brasil - CEP 18618-689
Jéssica Karina da Silva Pachú, Departamento de Entomologia e Acarologia - LEA Avenida Pádua Dias, 11 - CEP 13418-900 - Piracicaba/SP, Brasil
* Autor para correspondência: tati_cg04@hotmail.com
